Comment faire taire un gène ... l’interférence ARN
Une des plus récentes découvertes en biologie moléculaire est que les ARN peuvent réguler l’expression des gènes. Pendant des années, on leur attribuait deux fonctions principales :
- Les ARN messagers (ARNm) sont des intermédiaires monocaténaires (à un brin) indispensables entre l’ADN, porteur de l’information génétique, et les protéines, expression de cette information.
- Les ARN ribosomaux(ARNr) et de transfert (ARNt), quant à eux, ont des rôles de "décodeurs" et catalyseurs de cette synthèse protéique, de par leur structure. (cf. schéma général)
Ce tableau s’est quelque peu compliqué, quand des scientifiques ont découvert l’ARNi (interférence ARN), ou comment l’expression des gènes est bloquée par une molécule d’ARN bicaténaire (à deux brins). Depuis, l’ARNi est devenu un outil expérimental révolutionnaire pour comprendre le rôle des gènes. Ancien mécanisme de défense du génome, il est impliqué dans le blocage de la transcription (la synthèse d’ARNm à partir de l’ADN) et celui de la traduction (la "lecture" de l’ARNm en peptide).
La découverte
Le mécanisme de l’ARNi repose sur un principe technique simple : en injectant des copies multiples d’ARN à deux brins (double strand ou dsARN) dans le ver Cænorhabditis elegans on bloque le fonctionnement du gène dont la séquence est complémentaire de ce dsARN. Comme beaucoup de découvertes majeures, celle de l’ARNi repose sur un résultat expérimental inattendu.
À la fin des années 1980- début 1990, des biologistes en physiologie végétale, travaillant sur le pétunia, eurent la surprise de constater que l’introduction de copies multiples d’un gène, codant pour la couleur pourpre foncé, loin de renforcer la couleur des fleurs, provoquait l’apparition de taches blanches sur les pétales. D’une manière encore inconnue, les gènes introduits (les transgènes) étaient non seulement devenus muets, mais avaient aussi bloqué les propres gènes de la plante-hôte. Le même résultat était obtenu lorsque les plantes étaient infectées par un virus génétiquement modifié pour contenir seulement des fragments du gène.
Ces résultats ne furent compris que lors de la publication des travaux de Fire et Mello, en 1998 : les virus à ARN multiplient leur matériel génétique via l’intervention de dsARN et les nombreuses copies de transgènes provoquent également la formation de dsARN. Le phénomène d’ARNi est donc un ancien mécanisme de défense cellulaire, apparu bien avant la divergence vers-plantes, qui bloque toute possibilité de multiplier de façon anarchique un matériel génétique "déviant" ou d’origine virale, normalement absent de la cellule.
Comment ça marche ?
Une des premières observations qui ont permis de comprendre le mécanisme de l’ARNi, est que les plantes dont les gènes sont rendus silencieux produisent de petits ARN de 20 à 25 nucléotides, complémentaires des gènes bloqués. Ces ARN, plus petits que les ARNm et les ARN ribosomaux sont restés longtemps indétectables.
L’ARNi a été reconstituée in-vitro, à partir d’extraits cellulaires de Drosophile. Tout d’abord, de longs dsARN sont fragmentés en ARN plus petits avec une structure spécifique : deux chaînes de 21 nucléotides dont 19 appariés en double brin et 2 nucléotides non appariés aux extrémités. Ces petits fragments ont été baptisés "short interfering RNA"ou siARN ; même synthétisés artificiellement, ils sont capables de rendre des gènes silencieux. L’enzyme qui coupe les dsARN en siARN est appelée DICER. C’est une endonucléase de la famille des RNAses III, spécifique des dsARN.
Les fragments d’ARN obtenus forment alors un complexe avec des protéines nommées RISC.Là, les brins des siARN "sens", semblables à celui du gène cible, sont enlevés, laissant les brins "antisens", complémentaires. Ceux-ci guident un complexe protéique vers l’ARNm produit par le gène, provoquant sa destruction. Ainsi, le gène est transcrit mais non traduit, d’où son silence apparent.
Ceci explique l’échec des expériences de transgenèse chez les pétunias. Les deux brins d’un transgène peuvent être transcrits (au lieu d’un seul brin, comme l’est un gène normal), produisant un dsARN au lieu d’un ARNm. Celui-ci est alors coupé en plusieurs siARN qui bloquent le gène d’intérêt. Plus il y a de copies du gène, plus la cellule le fait "taire" !
L’ARNi ne fonctionne pas ainsi pour tous les organismes. Par exemple, une ARN polymérase dépendante d’un ARN peut intervenir : elle utilise le brin antisens d’un siARN comme amorce pour synthétiser plus de dsARN, amplifiant le mécanisme. Chez les plantes, ce phénomène se propage à travers les tissus non-reproductifs (somatiques), par transfert de dsARN de cellule à cellule, générant une extension de la résistance aux infections virales. Un tel mécanisme est peu probable chez la drosophile ou les vertébrés, car la polymérase en question n’a pas été trouvée chez eux.
voir l’animation : http://www.nature.com/focus/rnai/animations/animation/ui_main.swf
À quoi ça sert ?
Les petits ARN interférents ou siARN pourraient avoir un rôle dans la lutte contre les éléments transposables, les gènes répétés (y compris les transgènes) et les virus.
Les transposons, ces "gènes sauteurs" facétieux qui ont la fâcheuse manie de parasiter l’ADN en se dupliquant pour s’insérer n’importe où, représentent près de la moitié des séquences génétiques chez l’humain. Les "faire taire" est crucial pour la stabilité du génome ! Les vers possédant des gènes mutés, impliqués dans l’ARNi sont incapables de bloquer certains transposons dans les tissus germinaux (à l’origine des cellules reproductrices). Il en est de même pour les végétaux.
Ce mécanisme pourrait aussi contrôler le degré d’expression de gènes dans certains tissus. Chez la drosophile, par exemple, la quantité de copies d’un transgène peut faire basculer un résultat : un certain nombre de copies génère une forte expression du gène d’intérêt, mais à partir d’une concentration limite, son expression, au contraire, se réduit. Les protéines qui "emballent" l’ADN sont impliquées ; leur structure est modifiée pour stopper la transcription. Chez la levure, ce procédé, observé sur les éléments transposables, s’étend curieusement aux régions adjacentes.
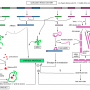
L’ARNi : siARN ou miARN ??? (voir schéma)
La révélation du rôle majeur des siARN a lancé les chercheurs dans une recherche frénétique de petits ARN. Selon la boutade d’un scientifique, on fait "les fonds de poubelle" : ce qui était considéré comme négligeable dans les fractions cellulaires est maintenant passé au crible. Le résultat a mené à la découverte, chez plusieurs espèces animales, de différents ARN de petite taille (19 à 25 nucléotides). Cette nouvelle classe a été nommée microARN ou miARN.
Ils sont codés par le génome, mais, contrairement aux ARN messagers, n’aboutissent pas à la production d’une protéine. En fait, ils régulent la production d’ARNm. Ils sont abondants, simple brin, et peuvent aller de quelques centaines à plusieurs milliers de molécules par cellule, soit 0,5 à 1% des gènes ; chez l’Homme, ils représentent 200 à 255 gènes. On pense qu’ils dérivent d’ARN double brin mais en "épingle à cheveu", c’est-à-dire un simple brin replié sur lui-même par complémentarité de bases. Ce qui fait que les miARN sont produits par le même mécanisme que celui qui coupe les dsARN en siARN, par DICER et autres enzymes. Leur mode d’action consiste à s’apparier partiellement avec un ARNm donné, bloquant ainsi sa traduction en protéine. La différence avec les siARN est qu’ils ne stoppent pas la transcription et l’ARNm n’est pas détruit. De plus, les siARN ciblent les gènes dont ils sont issus, tandis que les miARN, plus ou moins spécifiques, régulent plusieurs gènes, peut-être une centaine ou plus par miARN. Leur efficacité dépend aussi du nombre de molécules fixées sur chaque ARNm, qui contient en effet plusieurs sites de liaison à l’extrémité 3’.
Dans la plupart des cas, que ce soit chez les plantes ou mammifères, ils sont impliqués dans la régulation de facteurs de transcription, indispensables au développement. Une des régulations exercée par les miARN, chez les nématodes, détermine le passage d’un stade larvaire à l’autre durant le développement post-embryonnaire. Chez les plantes, les miARN agissent également sur des stades du développement ; chez la drosophile, ils contrôlent la division et la mort cellulaires. Chez l’Homme, leur découverte trop récente ne permet pas encore de déterminer leur rôle ; celui-ci est probablement majeur, puisque les gènes ont été conservés chez de nombreuses espèces au cours de l’Evolution.
L’intérêt de ces deux types moléculaires est que les siARN ciblent si précisément l’ARNm qu’ils ne peuvent intervenir par inadvertance sur n’importe quelle synthèse protéique, tandis que les miARN, eux, ne peuvent agir qu’en certaine concentration, ce qui suppose une régulation de leur expression.
Compliquons un peu...
D’autres mécanismes faisant intervenir de petits ARN sont peu à peu découverts. Certains siARN particuliers, trouvés chez le cilié Tetrahymena thermophila, sont capables de se fixer directement à l’ADN de manière spécifique et de provoquer sa fragmentation. Les deux gènes impliqués dans ce mécanisme d’ARNi ont été retrouvés chez d’autres espèces et sont encore à l’étude.
L’an dernier, une nouvelle classe d’ARN de 20 à 22 nucléotides a été décrite chez C.elegans : les "minuscules ARN non codants"(tiny non-coding RNAs) ou tncARN. N’ayant pas deux séquences complémentaires proches, ils ne sont donc pas en épingle à cheveux. Bien que produits par DICER, ils ne sont pas conservés chez toutes les espèces. Leur fonction n’est pas connue, mais beaucoup semblent complémentaires d’ARNm et pourraient aussi intervenir dans leur dégradation.
Enfin, cette année, un petit dsARN non codant, de 20 nucléotides, baptisé smARN (small modulatory RNA) a été isolé à partir de cellules souches de neurones, dans l’hippocampe d’une souris adulte. Il a la même séquence qu’un fragment d’ADN de 21 à 23 nucléotides qui inhibe l’expression de gènes spécifiques aux cellules nerveuses dans les autres types cellulaires ; il a été conservé au cours de l’Evolution chez les tous les vertébrés. Le smARN, dans les neurones, interagit avec les protéines régulatrices de ce gène en favorisant son expression et le développement neuronal. (cf.résumé dans le lexique*)
L’intérêt clinique
Les progrès récents de la génétique visent à identifier les anomalies responsables de maladies. Les effets obtenus par des mutations dirigées spécifiquement sur un gène permettent d’en comprendre le rôle, mais la technique se pratique surtout sur les organismes à reproduction rapide, comme la drosophile. Chez les mammifères, ce procédé long et coûteux pourrait être amélioré par l’emploi d’ARNi.
Cette technique est relativement simple chez C.elegans : on injecte les dsARN dans le ver, ou on le nourrit avec des bactéries qui les expriment, et l’on observe l’effet de l’inhibition du gène étudié. Sur les 19000 gènes de cet animal, on possède déjà une banque de 12000 dsARN. Ceci a permis de découvrir des gènes impliqués dans l’obésité ou le vieillissement cellulaire chez le ver et dans le métabolisme du cholestérol ou la formation du cœur chez la drosophile. Si ces gènes sont conservés dans l’Evolution, on comprend l’intérêt de leur étude pour l’espèce humaine !
Quelques essais in-vitro ont par ailleurs été menés chez l’Homme, mais les dsARN, de grande taille, tuent nos cellules, aussi la recherche emploie-t-elle de petits segments de 21 à 22 paires de bases. 8000 des 35000 gènes humains ont déjà révélé leur rôle, en particulier dans les cancers. Mais c’est aussi comme outils que les petits ARN pourraient se montrer redoutables par exemple en bloquant spécifiquement les gènes responsables de la formation de tumeurs, ou la propagation de virus. Des effets prometteurs ont déjà été obtenus chez le rat, en bloquant les virus de l’hépatite C et B, mais le problème reste celui du mode d’administration des petits dsARN : comment cibler l’organe concerné ?
Conservés au cours de l’Évolution, les petits ARN nous permettent de lutter contre des perturbations du génome ou des envahisseurs viraux, régulent l’expression de gènes majeurs tout au long du développement...on pense même qu’ils interviendraient dans les périodes de stress cellulaire, lors d’un changement radical de l’environnement. Des altérations de la transcription de l’ADN seraient "passées au crible"de ces régulateurs et sélectionnées si elles se révélaient avantageuses. Plus aucun doute, ces petites molécules vont sortir de l’ombre pour éclipser la "star ADN".
L.G
D’après Nature : vol.430- 8/7/2004 - C.D.Novina et P.A.Sharp : "The RNAi revolution".
(*)Lexique des petits ARN :
– siARN : short interfering RNAs : Une classe d’ARNs bicaténaires de 21 à 22 nucléotides de long, provenant de la dégradation de dsARN, ARN double-brin de plus grande taille. Les siARNs rendent les gènes silencieux en favorisant la dégradation des mARN auxquels ils se lient spécifiquement, ou en faisant intervenir des protéines inhibitrices sur l’ADN complémentaire de leurs séquences.
– miARNs : MicroARNs Un groupe de19 à 25 nucléotides, monocaténaires, codés par le génome et souvent conservés au cours de l’Evolution. Les miARNs interviennent dans le développement des organismes. Ils bloquent l’expression des gènes lors de la synthèse protéique.
– tncARNs : tiny non coding ARNs Un nouveau type de 20 à 22 nucléotides chez C.elegans. Ils ne sont pas conservés au cours de l’Evolution, mais sont régulés au cours du développement. Leur fonction reste inconnue.
smARNs : small modulatory RNAs Un petit ds ARN identifié chez la souris, qui permet l’expression de gènes spécifiques des neurones adultes.
Pour en savoir plus :
– ARNi : http://www.nature.com/focus/rnai
– Très belles animations en 3D sur la synthèse protéique et l’ARNi, avec lexique et possibilité de les télécharger :http://www.nature.com/focus/rnai/animations/
– Pour la science :
"Les ADN et les ARN antisens" - n°149- Mars 1990 ;
"La police du génome" n°312 - Octobre 2003, article très clair qui reprend les mêmes idées que l’article traduit ci-dessus sur les siARN et miARN, avec des exemples d’expériences. Les photos fig.3c et d correspondent à l’expérience de blocage du virus de l’hépatite C ( Nature 418, 38 - 39 - 04 July 2002"Gene expression : RNA interference in adult mice").
– Compléments :
Un schéma généralrésumant les divers rôles connus des ARN. Les "riboswiches", observés entre autres chez des bactéries, sont des ARN messagers particuliers qui, selon leur conformation dans l’espace, favorisent ou non leur traduction en protéine. Ils s’apparentent un peu, pour cela, aux "ribozymes" ou ARN à activité enzymatique, et régulent leur propre expression. L’exemple donné montre comment se fait la synthèse de vitamine B12 : en absence de celle-ci, la cellule transcrit et traduit un gène codant pour l’enzyme de synthèse de B12. En présence de la vitamine, ce n’est pas l’enzyme qui est inactivée ou le gène, mais l’ARNm qui change de conformation et devient inaccessible aux ribosomes. Une autre technique pour "éteindre" un gène, non représentée ici, consiste à utiliser un ARNm "antisens", complémentaire de l’ARNm visé.