Transcription et traduction en langage Python.

Utiliser l’algorithmique et le langage informatique Python afin de modéliser et automatiser la transcription et la traduction avec des élèves de première spécialité SVT lors d’une séance en classe inversée.
Contexte et objectifs :
– Lors de la séance précédente, le mécanisme de la transcription a été étudié.
– À la maison (séance inversée), il est demandé aux élèves :
* d’exploiter des ressources afin de déterminer le système de correspondance entre la séquence de nucléotides de l’ARNm et la séquence d’acides aminés d’une protéine.
* d’élaborer un algorigramme (= représentation graphique d’un algorithme ; il permet de représenter chaque opération élémentaire au moyen d’un symbole graphique) des mécanismes de transcription et de traduction.
– Retour en classe : Séance de cours de 2 heures en salle informatique équipée de 20 postes.
Les objectifs d’apprentissage sont :
* de consolider les connaissances acquises sur la transcription et le travail sur le code génétique réalisé à la maison pour transcrire et traduire une portion d’un gène.
* de montrer aux élèves l’intérêt d’utiliser un programme informatique pour automatiser la transcription et la traduction d’un gène constitué d’un millier de nucléotides.
C’est aussi l’occasion de leur montrer que les SVT, comme les mathématiques et la physique, ont besoin dans certains cas de la programmation et d’évoquer la filière bio-informatique peu connue des élèves.
| Compétences travaillées | Connaissances associées | Attitudes associées |
|---|---|---|
|
Thème 1 : La Terre, la vie et l’organisation du vivant – L’expression du patrimoine génétique La séquence de l’ADN, succession des quatre désoxyribonucléotides le long des brins de la molécule, est une information. Les molécules d’ARN sont synthétisées par complémentarité des nucléotides à partir de l’ADN lors d’un processus dénommé transcription. Les ARN messagers dirigent la synthèse de protéines lors d’un processus dénommé traduction. Le code génétique est un système de correspondance qui permet la traduction de l’ARN messager en protéines. L’information portée par une molécule d’ARN messager (le message génétique) est ainsi convertie en une information fonctionnelle (la séquence des acides aminés de la protéine).Appréhender la différence essentielle entre information et code. |
|
Outils numériques utilisés :
Pour la séance inversée : Les documents à exploiter sont en ligne sur un mur virtuel (Créer un mur collaboratif en classe avec padlet et Netboard, une alternative à Padlet.). Il est aussi possible de faire ce mur virtuel en utilisant l’ENT de votre établissement.
Pour la séance en classe, le logiciel de programmation choisi est Edupython 2.7 car c’est celui utilisé par les professeurs de mathématiques du lycée. Il ne nécessite pas d’installation, il suffit de télécharger le dossier zippé sur le site http://edupython.tuxfamily.org/, puis le décompresser.
Déroulé des étapes du travail et remarques diverses :
• Distribution de la consigne pour la séance à la maison (séance inversée) :
| Consigne |
|---|
| 1 .Les protéines sont constituées d’une séquence d’acides aminés. Il existe 20 acides aminés différents. Pour l’ADN et l’ARN, il n’existe que 4 variétés de nucléotides différents. Quelle est la correspondance entre la séquence des nucléotides de l’ARNm et la séquence des acides aminés de la protéine ? Exploiter les documents 1 et 2 présents sur le mur virtuel situé au lien suivant : https://padlet.com/profsvt/codegene... afin de déterminer le système de correspondance entre ARNm et protéine. Vous justifierez votre réponse en argumentant. 2. Répondre à la question posée dans le document 3. |
• Séance en classe :
La séance est divisée en plusieurs temps. Le gène TAS2R38 a été choisi car il sera de nouveau utilisé lors d’une prochaine séance.
– Premier temps : Transcription et traduction d’une portion du gène « à la main ».
- Consigne distribuée aux élèves :
Voici la portion codante de la séquence du gène TAS2R38 assurant la production de la protéine TAS2R38.
Cette protéine présente dans la membrane des cellules de la langue est un récepteur responsable de la sensibilité au goût amer des aliments.

En vous servant de votre travail maison sur le code génétique et de vos connaissances, transcrire (=passer de l’ADN à l’ARNm) et traduire (= passer de l’ARNm à la protéine) la portion de la séquence du gène TAS2R38 proposé ci-dessus afin d’obtenir la séquence d’acides aminés de la protéine TAS2R38.
Appeler le professeur pour validation.
– Second temps : Algorithmes et mise en activité sur Edupython
- Consigne distribuée aux élèves :
Mais vous n’aviez qu’une portion du gène... Il contient en réalité plus d’un millier de nucléotides (Cf. séquence vidéoprojetée) ! Vous pouvez bien sûr continuer à le transcrire et à le traduire à la main mais cela prendrait beaucoup de temps... Une solution, automatiser la transcription et la traduction grâce à un programme informatique qui vous permettra de transcrire et de traduire cette séquence en un temps record mais aussi toutes autres séquences que vous pourriez disposer !
D’ailleurs des étudiants en bio-informatique l’ont bien compris... Afin de faciliter leur travail, ils ont développé un programme informatique de transcription et de traduction en langage Python. Ce programme doit leur permettre, à partir de n’importe quelle séquence de nucléotides d’un gène d’obtenir la séquence d’acides aminés de la protéine correspondante. Mais, les étudiants ne sont pas d’accord entre eux...
a. Par groupe de 4, mettre en commun vos algorithmes de transcription et de traduction réalisés à la maison.
b. À partir des ressources à votre disposition (fiches techniques + votre réponse à la question 1), tester les programmes de chaque étudiant afin de déterminer celui ou ceux qui sont corrects ainsi que celui ou ceux qui ne le sont pas. Vous justifierez chacun de vos choix en argumentant.
Les programmes Python de chaque étudiant (transcription_traduction1, transcription_traduction2...) sont à ouvrir avec l’éditeur d’Edupython présent sur les postes informatique.
Coup de pouce : Pour comprendre chaque étape des programmes Python, copier/coller des parties de programme sur le site Python tutor présent au lien suivant : http://pythontutor.com/visualize.ht...
Appeler le professeur pour validation.
Voici les fiches techniques et les aides distribuées aux élèves :
- Fiche technique pour utiliser l’éditeur d’Edupython :
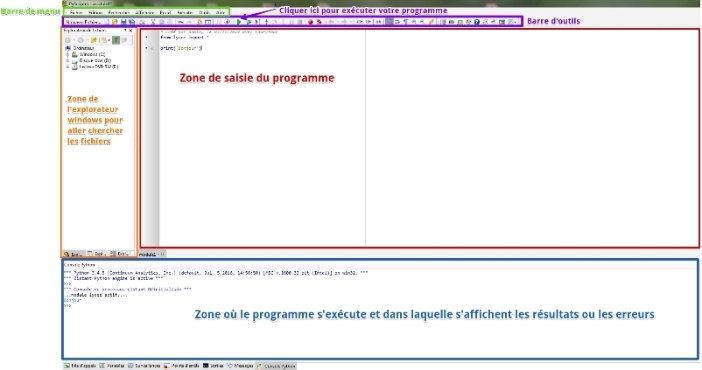
Fiche technique Edupython (version papier) :
- Aides pour utiliser le site Python tutor :
– Copier/coller les parties de programme dans la fenêtre de Python tutor afin d’en comprendre les actions.
– Cliquer sur « Visualize Execution ». Si un message en rouge apparaît, c’est soit qu’il y a une erreur dans le programme, soit que le programme est trop long. Si c’est le cas, corriger l’erreur là où elle est indiquée ou raccourcir le code en enlevant les explications notées après les #.
– Cliquer sur « Next » jusqu’à la fin du programme. Chaque action effectuée apparaît dans la fenêtre de droite. Vous pouvez revenir en arrière en cliquant sur « Prev » et modifier le code du programme en cliquant sur « Edit this code ».
- Fiches techniques d’instructions du langage python :
Elles sont aussi présentes sur le site d’Edupython.
Les bases du langage Python :Les instructions pour les chaînes de caractères :
- Voici les 5 programmes python mis à la disposition des élèves :
Programme Transcription-Traduction 1 :Programme Transcription-Traduction 2 :
Programme Transcription-Traduction 3 :
Programme Transcription-Traduction 4 :
Programme Transcription-Traduction 5 :
- Voici des exemples de traces écrites d’élèves :
Remarque : La rédaction de l’argumentaire n’était pas une compétence évaluée de façon formative durant cette séance.
Exemples de traces écrites d’élèves :
Une mise en commun en phase dialoguée a été réalisée en fin de séance.
– Activités complémentaires :
Les activités complémentaires présentées ci-dessous avaient été prévues pour les élèves les plus rapides ou les plus experts. Elles n’ont pas pu être réalisées durant cette séance. Elles se sont finalement déroulées lors de la séance réutilisant ce gène.
- Consigne distribuée aux élèves :
Activités complémentaires pour les plus rapides et/ou les plus experts :
– Déterminer la longueur des différentes séquences en utilisant la console python et l’instruction len.
– Le gène TAS2R38 existe sous 2 formes alléliques. Les 2 allèles diffèrent par 3 mutations par substitution (Cf. tableau ci-dessous). Les individus possédant 2 exemplaires de l’allèle muté sont insensibles au goût amer.
Créer votre propre programme sur Edupython en téléchargeant le fichier Mutations et l’ouvrir avec Edupython. Ce programme doit permettre de modifier la séquence d’ADN du gène (=mutations) ainsi que de déterminer les conséquences de ces 3 mutations sur la protéineTAS2R38.
Coup de pouce : Utiliser le fichier Mutations_secours si vous n’y arrivez pas.
Compléter ensuite le tableau ci-dessous avec les résultats.
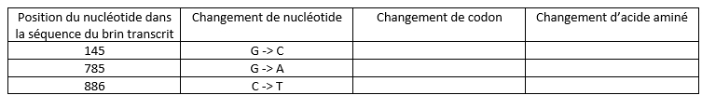
Voici les programmes python correspondant à ces activités :
Programme Mutations : Programme Mutations_Secours :
Correction du programme Mutations :
Voici le diaporama utilisé lors de la séance. Il contient aussi la correction des activités.
Diaporama de la séance :
Bilan :
– En amont de la séance, j’ai demandé aux élèves s’ils connaissaient l’algorithmique et la programmation puis le langage python. Certains élèves en faible nombre programment chez eux, mais n’utilisent pas python comme langage, la majorité ont déjà utilisé au moins une fois python en mathématiques. Ils étaient avertis que nous allions utiliser ce langage en SVT, ce qui a été l’occasion de leur parler de la bio-informatique.
– J’ai ainsi fait le choix de réaliser moi-même les programmes, car aucun élève ne connaissait ce langage. De plus, les erreurs créées dans les programmes sont des erreurs directement liées à la transcription et au code génétique, le but étant que les élèves approfondissent leur compréhension des mécanismes de transcription et de traduction et qu’ils manipulent leurs connaissances afin de les consolider.
– La séance s’est bien déroulée : les élèves ont joué le jeu et l’environnement Python ne leur a finalement pas posé problème même pour les novices. Certains élèves ont même modifié les programmes pour tester leur solution.
– Les activités complémentaires ont été réalisées lors d’une séance de TP, la création des mutations a posé plus de problèmes à certains élèves. Cependant une fois le principe de découpage en langage Python (slice) compris, la réalisation a été possible.
Pour information, j’ai commencé la programmation en langage python grâce à des MOOC délivrés par FUN. Voici les liens dans l’ordre où je les ai suivis :
– MOOC Python 3 : Des fondamentaux aux concepts avancés du langage
– MOOC S’initier à l’enseignement en Sciences Numériques et Technologie
– MOOC Bio-informatique : algorithmes et génomes
C’est d’ailleurs grâce à ce dernier que l’idée d’utiliser l’algorithmique et Python avec les élèves a commencé à germer dans mon esprit...
En espérant vous avoir donné envie d’essayer... À vous de jouer !







